Par Mariana Bego
De nouveaux variants du virus responsable de la COVID-19 sont signalés presque tous les jours. Puisqu’il est difficile de parvenir à un système de nomenclature consensuel, les chercheurs donnent leurs propres dénominations créatives aux variants afin de les distinguer les uns des autres. Le nom des variants a commencé à se diversifier dès qu’on s’est mis à remarquer leur variété à la fin de l’année dernière, ce qui a compliqué les choses. Pour ajouter à la confusion, les termes « variant », « lignée » et « souche » sont souvent utilisés de manière interchangeable.
Un système de nomenclature, qui repose sur la relation évolutive entre les lignées du SARS-CoV-2, a donné le nom de B.1.17 au variant décrit pour la première fois au Royaume-Uni, mais d’autres l’ont plutôt nommé 20I/501Y.V1 pour rappeler ses principales mutations. Ce variant est également connu sous le nom de VOC 202012/01, conformément aux recommandations de l’Organisation mondiale de la Santé (OMS). Constatant que ces dénominations alphanumériques prêtaient à confusion, un groupe de chercheurs inspirés l’a plutôt rebaptisé « Nelly »!
À titre d’exemple, décortiquons les origines des noms possibles de ce variant :
Nom selon le lieu et la date (le variant britannique ou VOC 202012/01) : Ce virus a d’abord été connu sous le nom de variant britannique du SARS-CoV-2. Puisque la nomenclature des systèmes de variants d’après la région géographique où ils sont signalés pour la première fois peut jeter un discrédit dangereux, l’OMS a eu vite fait de déconseiller cette nomenclature. En janvier 2021, elle a créé un nouveau système inspiré des concepts de variant à l’étude (Variant Under Investigation, ou VUI) et de variant inquiétant (Variant of Concern, ou VOC), suivi de l’année et du mois de sa découverte. Il est ainsi devenu le variant VOC 202012/01.
Découvrez-en davantage sur le sujet dans un récent éditorial de Nature, rédigé en anglais.
La nomenclature découlant de la mutation (N501Y et 20I/501Y.V1) : Certains noms proposés mettent en évidence des mutations importantes des variants d’intérêt. Ce variant a porté le nom préliminaire de N501Y pour refléter le fait que l’acide aminé en position 501 est passé d’une asparagine à une tyrosine (abrégés par N et Y, respectivement, conformément aux normes de nomenclature biochimique standards). Cependant, les variants transportent souvent plus d’une mutation au potentiel intéressant. Des chercheurs de Nextstrain ont tiré profit de cette information pour proposer de nommer les variants d’intérêt d’après les constellations de mutations dont ils sont porteurs. Nextstrain est un projet de logiciel libre qui partage des données sur les génomes des agents pathogènes, conjointement avec de puissants outils d’analyse et de visualisation. Ce sont eux qui ont donné le nom de 20I/501Y.V1 à ce variant. Des variants distincts porteurs d’ensembles de mutations importantes recevraient des noms semblables.
De nombreux chercheurs souhaitent éviter les noms tirés des mutations individuelles. Ce nom permet bel et bien aux chercheurs d’associer les variants aux mutations susceptibles d’influencer leurs comportements, mais il ne tient aucun compte d’autres changements importants du variant.
Pour en savoir plus, consultez le site Web de Nextstrain, en anglais.
Le système Pango (B.1.1.7) : Rambaut et ses collègues ont d’abord proposé de nommer les variants d’après une nomenclature dynamique qui produit des désignations techniques comme B.1.1.7. Pour être valide et largement acceptée, cette désignation doit a) rendre compte de la diversité génétique locale et mondiale; b) retracer les lignées émergentes; c) être à la fois solide et souple pour intégrer la diversité à mesure qu’elle se manifeste et d) demeurer dynamique au fil du temps. Ils proposent d’utiliser le terme
« lignées » (plutôt que « clades » ou « génotypes ») pour le virus SARS-CoV-2, parce qu’il rend bien sa nature dynamique.
Un arbre phylogénétique est un diagramme qui représente les relations évolutives entre les organismes, dans le cas qui nous intéresse les virus SARS-CoV-2 en circulation. Les chercheurs proposent de cartographier chaque virus séquencé dans un arbre phylogénétique et de faire commencer le nom de chaque lignée majeure (ou branche d’arbre) par une lettre (voir la figure 1). Les deux branches principales sont des virus de la lignée A, représentée par Wuhan/WH04/2020, et de la lignée B, représentée par Wuhan-Hu-1, dont semblent dériver la plupart des cas humains enregistrés jusqu’à présent. Rambaut et ses collègues ont nommé leur système de nomenclature Pango (du verbe latin pango à la première personne, qui signifie « je répare », « je fixe », « je stipule »). Un récent addenda au manuscrit original, publié à la fin de janvier 2021, contient des liens vers plusieurs outils en ligne pour classer les séquences du génome du SARS-CoV-2.
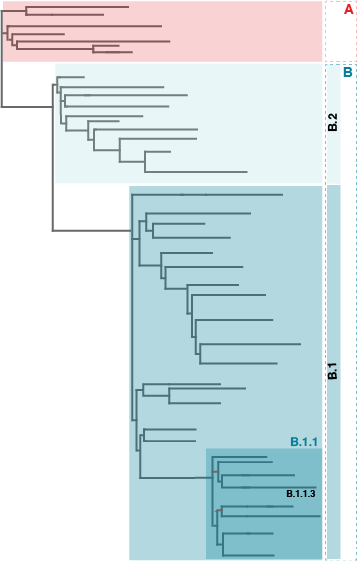
Figure 1: Arbre phylogénétique utilisé pour dériver les noms utilisés dans la nomenclature des lignées Pango.
Vous pouvez lire leur article ici, en anglais.
La nomenclature inspirée des ouragans (Nelly) : Áine O’Toole, une biologiste évolutive de l’Université d’Édimbourg, a présenté de nouveaux noms dans une entrevue récente au Guardian, car les précédents lui semblaient « des noms à coucher dehors ». Selon son système, N501Y est devenu « Nelly », D614G, « Doug », et E484K, « Eeek ». « À l’origine, nous devions l’appeler Erik, mais nous travaillons beaucoup avec un Eric et ne voulions pas vraiment conjuguer le nom de cette mutation avec celui d’une personne que nous connaissions », relate-t-elle.
Pour éviter de froisser leurs amis et les membres de leur famille, les auteurs de la prépublication dont il est question dans le blogue suivant ont sélectionné des noms d’animaux pour leur système de nomenclature provisoire. Le nom du variant clé décrit est donc passé des nomenclatures des lignées Pango inspirées par les arbres phylogénétiques à des noms d’oiseaux. Par exemple, B.1.2 est maintenant devenu Robin, B.1.234, Mockingbird, et B.1.1.220, Bluebird. Il y a aussi les variants Pelican, Yellowhammer et Quail. D’après les auteurs du manuscrit, les surnoms peuvent fonctionner à court terme, mais ils ne représentent guère une solution à long terme. « Et si on ajoute tous ces oiseaux, on se retrouvera bientôt avec un zoo! », a fait remarquer à The Guardian l’auteure principale de ce manuscrit, Emma Hodcroft, qui est également membre de Nextstrain.
Ceci est lien vers l’article publié la semaine dernière en anglais dans The Guardian.

